A Pfizer/BioNTech és a Moderna mRNS-vakcinák részletes elemzése és kritikus értékelése
Ez a cikk a https://www.ncbi.nlm.nih.gov/labs/pmc/articles/PMC8310186/ fordítása.
Szerző: Xuhua Xia Hatem
A. Elshabrawy, tudományos szerkesztő
Összefoglaló:
A Pfizer/BioNTech és a Moderna mRNS-vakcinák tervezésekor számos, különböző típusú optimalizálást végeztek. Ezt azért tették, mert vakcinában lévő mRNS megfelelő optimalizálása csökkentheti az egyes injekciókhoz szükséges dózist, ami hatékonyabb immunizációs programokat eredményezhet. A vakcina mRNS-komponenseinek rendelkezniük kell 5′-UTR-rel, hogy a transzláció beindításához a mRNS-hez a riboszómák hatékonyan tudjanak kapcsolódni, a transzláció hatékony meghosszabbodásához pedig optimális kodonok használatára van szükség, a transzláció hatékony befejezéséhez pedig optimális stop-kodonra. Mind az 5′-UTR-nek, mind a downstream 3′-UTR-nek az mRNS stabilitása érdekében optimalizáltnak kell lennie. Mindkét oltás esetében az uridin N1-metil-pszeudourininnel (Ψ) való helyettesítése megnehezíti ezen optimalizálási folyamatok némelyikét, mivel a Ψ könnyedén hoz létre bázispárosodást két nukleotid között az RNS-molekulákban, mint az U. A különböző optimalizálások ütközhetnek egymással, így kompromisszumokra lehet szükség. Kiemelem a Pfizer/BioNTech és a Moderna mRNS-vakcinák közötti hasonlóságokat és különbségeket, és kitérek az egyes vakcinák előnyeire és hátrányaira, hogy megkönnyítsem a jövőbeli vakcinafejlesztést. Ezen belül rámutatok néhány nem megfelelően elvégzett optimalizálásra a két mRNS-vakcina tervezésében.
Kulcsszavak: SARS-CoV-2, mRNS-vakcina, a transzláció beindítása, kodonoptimalizálás, transzlációs termináció, RNS szekunder struktúra, RNS-stabilitás.
1. Bevezetés
A két leggyakrabban használt SARS-CoV-2 vakcina, a Pfizer/BioNTech [1] és a Moderna [2] által gyártott vakcina, mindkettő mRNS-vakcina. A Pfizer/BioNTech BNT-162b2 szekvenciája nyilvánosan hozzáférhető [3], a Moderna mRNS-1273 szekvenciáját pedig nemrégiben szekvenálták [4]. Mindkét mRNS ugyanazt az S-2P fehérjét kódolja [5,6], amely a referencia SARS-CoV-2 genomban (NC_045512) található spike fehérjétől két aminosavval különbözik, azaz a 986-os és 987-es helyen lévő KV aminosavakat PP-vel helyettesítették, hogy a keletkező spike fehérjét prefúziós állapotban stabilizálják, hogy a gazdaszervezet immunrendszerét a vírus gazdasejtbe való belépése előtt felkészítsék a vírus felismerésére [7,8]. Bár a Pfizer/BioNTech BNT162b2 mRNS-ének és a Moderna mRNS-1273-nak ugyanaz az aminosav-szekvenciája, számos más tekintetben különböznek, például az 5′-UTR kialakításában, a kodonoptimalizálásban és a 3′-UTR-ben. Jellemzően a transzláció iniciálása a döntő lépés a transzlációban, és hatékonysága nagymértékben függ attól, hogy az 5′-UTR milyen gyorsan képes a riboszómát az mRNS-re telepíteni [9]. A transzlációs elongáció akkor válik korlátozóvá, ha a transzlációs iniciáció nagy hatékonyságú [10,11]. A vakcina mRNS hatékony transzlációra való optimalizálásával csökkenthető a gazdasejtekbe juttatandó oltóanyag mRNS példányok száma.
Mivel az emlős gazdasejtek megtámadják a nem módosított exogén RNS-t [12,13], az összes U nukleotidot N1-metil-pszeudouridinre (Ψ) cserélték [14,15]. A Ψ azonban a bázispárosodásban jobban ingadozik, mint az U, és nemcsak A-val és G-vel, hanem kisebb mértékben C-vel és U-val is képes párosodni [16]. Ez valószínűleg növeli a kodon téves olvasását egy közel-kognitív tRNS által. Amikor a stop-kodonokban az U nukleotidot Ψ-re cserélték, megnőtt a stop-kodonok közel-kognitív tRNS-ek általi félreolvasásának aránya [17]. Az ilyen átolvasási események nemcsak az immunogén fehérjék számát csökkenthetik, hanem egy hosszabb, ismeretlen sorsú, potenciálisan káros hatású fehérjét is létrehozhatnak.
Alaposan megvizsgáltam és kritikusan értékeltem a Pfizer/BioNTech és a Moderna által gyártott vakcina mRNS különböző optimalizálási stratégiáit az 5′-UTR-től a 3′-UTR-ig, kiemelve azok hasonlóságát és különbségeit. Genomikai és transzkriptomikai adatok felhasználásával rámutattam néhány, nem megfelelően elvégzett optimalizálásra a két mRNS-vakcina kialakítása során. A cél az, hogy a lehető legjobban elősegítsük a jövőben a jobb stratégiák kidolgozását a vakcina mRNS optimalizálásában.
2. Anyagok és módszerek
2.1. Szekvenciaadatok
Az mRNS optimalizálás túlnyomó részben a magasan expresszált fehérjekódoló gének és az átlagos fehérjekódoló gének közötti kontraszton alapul. A cikkben a riboszómális fehérjék génjeit használtuk a funkcionálisan fontos és magasan expresszálódó gének képviselőjeként. A HUGO génnómenklatúra bizottság (HGNC a https://www.genenames.org/ oldalon, elérés: 2021. június 20.) 35 kis és 54 nagy riboszómális fehérje gént tüntet fel. Ezeket a géneket ezután a www.ncbi.nlm.nih.gov/gene (hozzáférés: 2021. június 20.) oldalról is le kellett tölteni. A letöltött gének közül néhány pszeudogén, pl. az rpL21 és két rpL7a izoforma. Az RPS4Y2 szintén pszeudogénként van jelölve az NC_000024-ben. Ezeket a géneket, valamint néhány más, a riboszómális fehérje génekkel homológ, de a legtöbb szövetben nem expresszálódó gént (RPL10L, RPL39L és RPL3L) kizártuk. Csak az ubiquitikusan expresszálódó riboszómális fehérje gének (33 RPS és 50 RPL gén) kerültek bevonásra. A FASTA formátumú kiegészítő szekvenciafájl (RP_Longest_isoform_ubiquitous.fas) tartalmazza az egyes riboszómális fehérje gének leghosszabb splice izoformáját. A tanulmányban szereplő egyes eredmények az összes splice izoformát tartalmazzák, így a kódoló szekvenciák (CDS-ek) teljes száma nagyobb, mint 83.
Az emberi genomokat (az 1-22. kromoszómák, X és Y (NC_000001-NC_000024) az NCBI-ból töltöttük le. Az 1. kromoszóma (Chr01) 11 327 annotált CDS-ét (beleértve a splicing izoformákat is) használtuk a humán gének reprezentatív készleteként, hogy szembeállítsuk a riboszómális fehérje génekkel (mint a magasan expresszált gének reprezentatív készletével). A 18-22. kromoszómák összes intronjának nukleotidfrekvenciáját (0,2640, 0,2178, 0,2262 és 0,2920 az A, C, G és T esetében) a háttérfrekvenciák helyettesítőjeként használtuk a pozíciós súlymátrix kiszámításakor.
A SARS-CoV és a SARS-CoV-2 (NC_004718 és NC_045512) referencia genomjait a GenBankból töltöttük le, akárcsak más, denevérekből izolált, rokon koronavírusokét (MN996532, MG772933, MG772934). A pangolinból izolált koronavírus szekvenciáját (pangolin|EPI_ISL_410721|2019) a GISAID-ból töltöttük le. A BNT-162b2 vakcina mRNS-e nyilvánosan hozzáférhető [3]. Az mRNS-1273 szekvenciáját Jeong és munkatársaitól [4] vettük át, és azt hipotatívnak kell tekinteni. Ennek a feltételezett szekvenciának az 5′-UTR szekvenciája azonban megegyezik a Moderna szabadalmában [18] szereplő egyik 5′-UTR szekvenciával (SEQ ID NO 181). Hasonlóképpen, e szekvencia 3′-UTR szekvenciája megegyezik egy másik Moderna szabadalomban [19] szereplő egyik 3′-UTR szekvenciával (SEQ ID NO 21), kivéve, hogy a hetedik triplet AUG helyett UAG a feltételezett szekvenciában. Ebben az összefüggésben biztonsággal feltételezhető, hogy a feltételezett szekvencia a valódi mRNS-1273.
2.2. Szövetspecifikus génexpresszió
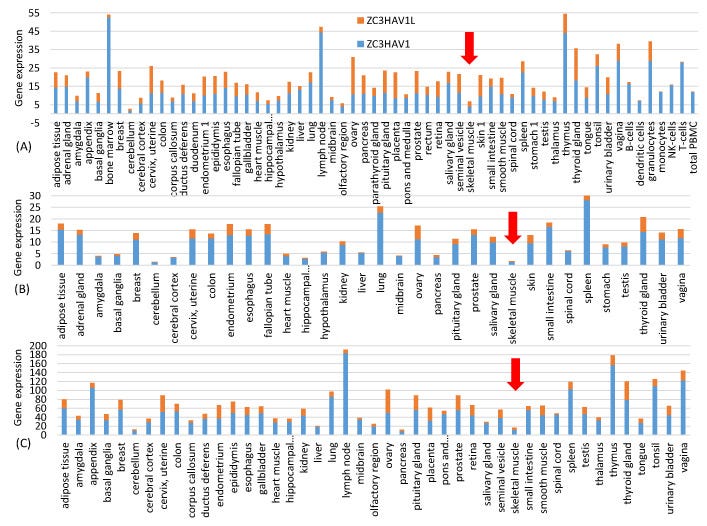
Mivel a két mRNS-vakcinát izombefecskendezéssel adják be, fontos a magasan expresszálódó izomgének jellemzőinek jellemzése. A Human Protein Atlas (www.proteinatlas.org, hozzáférés: 2021. június 20.) szövetspecifikus génexpressziós adatokat tartalmaz. A honlapon található rna_tissue_consensus.tsv.zip fájl 62 szövet génexpressziós adatait tartalmazza. A vázizomzatban 19 670 emberi fehérjekódoló gén (köztük 13 mitokondriális fehérjekódoló gén) génexpresszióját jellemezték. A géneket a kifejeződésük alapján rendeztem, és a vázizomban magasan kifejeződő reprezentatív géneknek a top 50-et választottam. Mellékeltem egy kiegészítő fájlt HEG50_Muscle.fas, amely tartalmazza ezen 50 magasan expresszált vázizomgén leghosszabb izoformájának kódoló szekvenciáit. Az 1. ábra adatai a szövetspecifikus génexpresszió három független számszerűsítéséből származnak, amelyeket a Human Protein Atlas három fájlja képvisel: proteinatlas.tsv, rna_tissue_gtex.tsv és rna_tissue_fantom.tsv. A zinc finger antivirális fehérje ZAP (NC3HAV1 és hosszú formája ZC3HAV1L) szövetspecifikus expresszióját mindhárom fájlból kivontuk és ábrázoltuk az 1. ábrán. A ZAP szinte teljes hiánya az izomsejtekben arra utal, hogy az izomsejtekbe injektált vakcinában lévő tüske mRNS-ek nem lesznek kitéve a ZAP által közvetített RNS-lebontásnak.
2.3. Szekvencia összeállítása és elemzése
A DAMBE [20] programot használtam a kódoló szekvenciák, a stop kodonok, valamint a kódoló szekvenciák előtti és utáni szekvenciák kinyerésére. A DAMBE-ből kiszámítottam a kodonfrekvenciákat, a kodonadaptációs indexet [21,22], a fordítási hatékonyság indexét (ITE) [11], a pozíció súlymátrixot (PWM) [23,24] és a minimális hajtási energiát (MFE) is. Az MFE kiszámítása a DAMBE-ben a Vienna RNS fold library [25] függvényeit használja.
2.4. Vírusos szubgenomiális mRNS a transzkriptomi adatokból
Tekintettel arra, hogy a SARS-CoV-2 vírusok által természetes úton átírt mRNS-szekvenciák potenciálisan fényt deríthetnek a vakcina mRNS optimalizálására, letöltöttem a SARS-CoV-2 transzkriptomikai adatokat [26] az NCBI SRA adatbázisából. A transzkriptomikai adathalmaz jó minőségű mintákat (pl. a SAR SRR11886744.sra fájlnak megfelelő GC-26/66) és rossz minőségű mintákat (pl. a SRR11886743.sra fájlnak megfelelő GC-55/68) tartalmaz. Letöltöttem az SRR11886744.sra fájlt, és elemeztem a proliferáló SARS-CoV-2 által természetesen termelt tüskefehérje szubgenomi mRNS-ét. Az elemzésből a természetesen termelt tüske mRNS 5′-UTR-jét vezettem le.
3. Eredmények és vita
3.1. Kodonoptimalizálás a transzlációs elongációs hatékonyság érdekében
A kodonoptimalizálásnak két szintje van. Az első az összetett kodoncsaládokat érinti. Például a SARS-2-S a referencia genomban (NC_045512) 42 Arg-maradékot tartalmaz, amelyből 30-at AGR-kódonok kódolnak, és csak 12-t CGN-kódonok (SRef oszlop az 1. táblázatban). A CGN-kódonok ilyen mértékű kerülése evolúciós szempontból ésszerű, mivel a gazdaszervezet zinc finger antivirális fehérjéi (ZAP, génnév ZC3HAV1) a vírus RNS-ben lévő CpG-dinukleotidokat célozzák meg, és a vírus RNS-genom lebontására sejtes RNS-degradációs komplexeket toboroznak [27,28,29]. A humán gének azonban gyakrabban használnak CGN, mint AGR kodonokat az Arg kódolásához. A magas expressziójúnak ismert riboszómális fehérje gének (33 RPS és 50 RPL) közül az Arg-maradékok 64,2%-át CGN kodonok kódolják. A BNT-162b2 és az mRNS-1273 8, illetve 28 AGR-kódonnal csökkentette az AGR-kódonok számát, a CGN-kódonok számának megfelelő növekedése mellett.

Felmerülhet a kérdés, hogy a CpG-dinukleotidok számának növekedése a vakcina mRNS gyors lebomlásához vezet-e, miután a ZAP által közvetített RNS-lebontási útvonalon keresztül bejutott a gazdasejtbe [27,28,29]. Az intramuszkuláris injekció esetében ez nem jelent aggodalmat, mivel a Human Protein Atlas három génexpressziós adatsorának megfelelően, amely a http://www.proteinatlas.org oldalon található (elérés: 2021. június 20.) [30], a ZAP szinte teljesen hiányzik a vázizomzatból (1. ábra). Ez rávilágít az mRNS-vakcinák egyik előnyére, mivel a vakcina beadására számos különböző, de kényelmes útvonal áll rendelkezésre, beleértve a szubkután, intramuszkuláris, intradermális, intratracheális, intravénás és intraperitoneális útvonalakat [31]. A vakcina mRNS-ében lévő magas CpG két további előnyt biztosít. Először is, a GC-gazdag mRNS-ek általában stabilabbak, mint az AU-gazdag mRNS-ek [32]. Másodszor, abban a valószínűtlen esetben, ha a vakcina mRNS-ek rekombinálódnának egy SARS-CoV-2 vírusba, az eredmény nem egy optimalizált spike fehérje génnel rendelkező vírus lenne, hanem egy CpG-gazdag RNS szegmens, amelyet a gazdaszervezet ZAP célzottan lebontana.
A Leu összetett kodoncsaládja hasonlóan optimalizált. A magasan expresszálódó humán riboszómális fehérje gének 81%-ban Leu-t kódolnak CUN kodonokkal. Emiatt a Leu majdnem minden UUR-kódját CUN-kódokká kódolták át mindkét vakcina mRNS-ben (1. táblázat). A Ser összetett kodoncsaládja új fordulatot hoz. Mindkét kodon alcsaládot nagyjából egyformán használják a Ser kódolására. Az AGY alcsaládot azonban könnyebb optimalizálni, mivel az AGC egyértelműen az AGU-val szemben az AGC a preferált kodon. Megjegyzendő, hogy a mutációs torzítás az U végződésű kodonoknak kedvezne, mivel az U gyakorisága az intronokban nagyobb, mint a C-é (0,2920 az U és 0,2178 a C esetében, 1. táblázat), de a magasan expresszálódó riboszómafehérje-gének az AGC-t részesítik előnyben az AGU-val szemben (144 az AGC és 95 az AGU esetében, 1. táblázat). Ezzel szemben az UCN alcsaládban az UCC és az UCU is gyakran használatos. Emiatt sok UCN Ser-kódot AGC-re kódoltak át a vakcina mRNS-ekben, különösen az mRNS-1273-ban (1. táblázat).
A kodonoptimalizálás második szintje a családon belüli optimalizálás. Két stratégiát alkalmaztak. Az első, amelyet a továbbiakban fundamentalista stratégiának nevezünk, egyszerűen az összes kodont a fő kodonnal helyettesíti. Az, hogy melyik kodon a fő kodon, koncepcionálisan két kritériumtól függ: (1) a nagymértékben kifejeződő gének előnyben részesítik a kodont, és (2) a legnagyobb mennyiségben előforduló tRNS dekódolja. Azonban e két kritérium hanyag alkalmazása tévedésekhez vezethet. Példaként a CGN kodoncsaládot veszem az Arg esetében, hogy bemutassam a két mRNS-vakcina hibás. optimalizálását.
Mindkét vakcina tervezői a CGN kodoncsaládban a CGG-t tekintették optimális kodonnak, és szinte az összes CGN kodont átkódolták CGG-re. A CGG mint optimális kodon kiválasztása látszólag a fenti két kritérium alkalmazásából adódott. Először is, a kodonhasználatról szóló EMBOSS [33] összeállítás, amelyet gyakran használnak a kodonoptimalizálás során, azt mutatja, hogy a CGG-t valamivel gyakrabban használják, mint a CGC-t. Másodszor, úgy tűnik, hogy a CGG-t több tRNS dekódolja, mint a többi szinonim CGN kodont. Az emberi genom hét tRNAArg/ACG gént tartalmaz a CGY kodonok dekódolására (ahol az ACG az antikodon, ahol az A inozin I-é deaminálódott), négy tRNAArg/CCG gént a CGG kodonok dekódolására és hat tRNAArg/UCG gént a CGA és CGG dekódolására (a harmadik kodonhelyen történő wobble pairing révén). Feltételezve, hogy a tRNS-bőség jól korrelál a tRNS-gének másolati számával, ami igaz a Saccharomyces cerevisiae-re [34], de más eukariótákra nem ismert, arra lehet következtetni, hogy a CGG-t több tRNS-gén fordítja (négy tRNAArg/CCG gén hat tRNAArg/UCG gén), mint a többi kodont, és ezért a két kritérium alapján ez a fő kodon. A két vakcina szinte az összes CGN kodont átkódolta CGG-re (1. táblázat).
A fenti, a tRNS-gén kópiaszámával kapcsolatos érvelés problematikus. Az emberi tRNS-gének közel fele nem expresszálódik [35], így nem használhatjuk a tRNS-gén kópiaszámát a sejtek tRNS-állományában lévő tRNS mennyiségének helyettesítőjeként. Emiatt a magasan expresszált gének által az alacsonyan expresszált génekhez viszonyított kodonpreferencia jobb működési kritérium a kodonoptimalizáláshoz. Az EMBOSS-ban [33] a humán gének kodon-összeállítása 1993-ban és 1994-ben készült, és nem volt cél, hogy csak a magasan expresszáltakat tartalmazza, így a CGG CGC-nél kissé magasabb használata egyszerűen mutációs torzításból adódhat (a G nukleotid gyakorisága következetesen magasabb, mint a C-é a humán intronokban).
Két bizonyíték is arra utal, hogy a CGG nem az optimális kodon. Az első a humán riboszómális fehérje gének ("RP" az 1. táblázatban) kodonhasználatát érinti, amelyekről ismert, hogy nagymértékben expresszálódnak. Ezek a gének a CGC kodonokat részesítik előnyben (1. táblázat). A második és közvetlenebb bizonyíték a vázizomsejtekben nagymértékben kifejeződő gének kodonhasználatából származik (amelyek itt azért relevánsak, mert a vakcina mRNS-t a lipid nanorészecskék injektálják és viszik a vázizomsejtekbe, hogy lefordítsák, bár a vakcina mRNS-t más szövetekbe is eljuthat). A vázizomzatban leginkább expresszálódó 50 gént a Human Protein Atlas konszenzusos expressziós adatkészletéből választottam ki a http://www.proteinatlas.org oldalon (hozzáférés: 2021. június 20.) [30], de kizártam azokat, amelyek CDS-ei 300 kodonnál kevesebbet tartalmaznak. A fennmaradó 26 gén (2. táblázat), köztük a leginkább izomspecifikus gének, mint a titin (TTN), az aktin (ACTA1) és a miozin (MYH1), szignifikánsan több CGC kodont használnak, mint CGG kodont (páros mintás t-próba, t = 3,075, DF = 25, p = 0,0034, 2-tailed teszt). Ezért a riboszómális fehérje gének által preferált CGC kodonokat a magasan expresszálódó izomgének is preferálják. További magasan expresszálódó fehérjekódoló gének a humán elongációs faktor 1α két izoformája (hEF1A1 és hEF1A2) és a poli(A)-kötő fehérje (hPABPC1). Ezek is több CGC-t használnak, mint CGG-t (a CGC:CGG arány 3:0 a hEF1A1 esetében, 8:6 a hEF1A2 esetében és 14:4 a hPABPC1 esetében).
Ezek a bizonyítékok arra utalnak, hogy a CGC jobb kodon, mint a CGG. Az mRNS-vakcinák tervezői (különösen az mRNS-1273, 1. táblázat) egy rossz kodont választottak optimális kodonnak.

Más kodoncsaládok optimalizálása egyszerű. A 2-szeres R végződésű kodonok esetében a háttérmutációs torzítás, amint azt az intronok nukleotidfrekvenciája tükrözi az emberi genomban, az A- végződésű kodonokat részesíti előnyben, de a riboszómális fehérje gének következetesen a G végződésű kodonokat részesítik előnyben minden 2-szeres R végződésű kodoncsaládban. Következésképpen a két mRNS-vakcinában a G végű kodonokat vették optimális kodonnak (3. táblázat a Glu-t kódoló GAR-kodonok esetében). A 2-szeres Y végződésű kodonok esetében a háttérmutáció az U végződésű kodonokat részesíti előnyben, de a riboszómális fehérje gének a C végződésű kodonokat részesítik előnyben, így a C végződésű kodon az optimális kodon. Van egy másik oka is annak, hogy az U végződésű kodonokat C végződésű kodonokká kódolják át. A két mRNS-vakcinában az összes U-nukleotidot N1-metil-pszeudouridinnel (Ψ) helyettesítették, amely minden mRNS nukleotiddal képes bázispárt alkotni, és ezért nem szabadna 2-szeres kodoncsaládokban használni. Például az Asp-ot kódoló GAΨ párosodhat a tRNAGlu antikodonjával, ami nemszinonim helyettesítésekhez vezethet. A C végű kodonoknál ez a probléma nem áll fenn, ami egy újabb ok arra, hogy az U végű kodonokat C végű kodonokká kódoljuk át.

A kodonoptimalizálás második stratégiája, amelyet a továbbiakban liberális stratégiának nevezünk, egyszerűen a fundamentalista stratégia kevésbé szélsőséges változata, amely minden szinonim kodont az optimális kodonnal helyettesít. Tegyük fel, hogy egy NNR szinonim kodoncsaládot a tRNS-1 által dekódolt NNG-vel és a tRNS-2 által dekódolt NNA-val. Tegyük fel továbbá, hogy a tRNS-1 kétszer olyan gyakori, mint a tRNS-2, és hogy a magasan expresszálódó gének az NNG kodont részesítik előnyben az NNA kodonnal szemben. A fundamentalista stratégia az összes kodont NNG-vel helyettesíti. A liberális stratégia a következő logikán alapul. Amikor egy sejt tele van NNG kodonokkal rendelkező mRNS-sel, a tRNS-1 olyan nagy igénybevételnek lesz kitéve, hogy kevésbé lesz elérhető, mint a tRNS-2, annak ellenére, hogy kétszer annyi tRNS-1 van a sejtben, mint tRNS-2. Emiatt optimálisabb lehet, ha néhány kódot a tRNS-2 által dekódolva tartunk.
E két stratégiát példázza a Glu-t kódoló GAR-kodonokat érintő kodonoptimalizálás (3. táblázat). A SARS-CoV-2 referencia genom (NC_045512) 34 GAA-kódont és 14 GAG-kódont tartalmaz a spike protein génjében. A Moderna mRNS-1273 a fundamentalista stratégiát követte, és az összes GAA kodont GAG-ra cserélte. Ezzel szemben a Pfizer/BioNTech BNT-162b2-je a liberális stratégiát követte, és 14 GAA-kódont változatlanul hagyott (3. táblázat). A Moderna következetesen a fundamentalista stratégiát alkalmazta az mRNS-1273 valamennyi kodoncsaládjára, míg a Pfizer/BioNTech következetesen a liberális stratégiát alkalmazta a BNT-162b2 kodonoptimalizálásában. Nincs szisztematikus értékelés e két kodonoptimalizálási stratégiáról a transzlációs hatékonyság tekintetében. Tekintettel a dózisbeli különbségre (100 μg az mRNS-1273 és 30 μg a BNT-162b2 esetében) és a hatékonyság egyenértékűségére, feltételezhető, hogy a Pfizer/BioNTech vagy a Moderna vakcina beadása ugyanannyi kódolt tüskefehérjét eredményez. Ez azt jelentené, hogy a Pfizer/BioNTech vakcinában lévő mRNS átlagosan valószínűleg körülbelül 3,3-szor annyi fehérjét termel, mint a Moderna vakcinában lévő mRNS.
A BNT-162b2-re és az mRNS-1273-ra alkalmazott kodonoptimalizálás a két vakcina mRNS esetében sokkal magasabb kodonadaptációs indexet (CAI) [21,22] és transzlációs hatékonysági indexet (ITE) [11,36] eredményez. A természetes koronavírusok S génjének CAI-ja < 0,7="" a="" tüskefehérje="" cds-ére,="" de="" a="" két="" kodonoptimalizált="" tüske="" cds-ének="" cai-ja="" 0,94925,="" illetve="" 0,97939="" (4.="" táblázat).="" az="" ite="" egy="" általánosított="" cai,="" amely="" figyelembe="" veszi="" a="" háttérmutációs="" torzítást="" [11].="" ennek="" értékei="" hasonlóan="" sokkal="" magasabbak="" a="" két="" vakcina="" mrns="" esetében,="" mint="" a="" természetes="" vírusoknál.="" a="" maximális="" cai="" és="" ite="" értékek="">

(1) A DAMBE [20] segítségével kiszámítva, a homo_sapiens_HEG_RibosomalProteins" kodonhasználati táblázattal..
A BNT-162b2 kisebb CAI- és ITE-értékei, mint az mRNS-1273 esetében, azt a benyomást kelthetik, hogy a BNT-162b2 kevésbé kodon-optimalizált, mint az mRNS-1273. Ez nem feltétlenül igaz. Mint már említettem, az mRNS-1273 kodon-optimalizálása a fundamentalista stratégiával történt (azaz minden vagy majdnem minden szinonim kodont az optimális kodonnal helyettesítettünk), míg a BNT-162b2-t a liberális stratégiával optimalizáltuk, amely kevésbé szélsőséges, mint az előbbi. A fundamentalista stratégia szükségszerűen magasabb CAI- vagy ITE-értékeket eredményez, mint a liberális stratégia. A liberális stratégia azonban hatékonyabb transzlációs elongációhoz vezethet, ha túl sok olyan kodon van, amely a legbőségesebb tRNS-t követeli, ahogyan azt korábban tárgyaltam.
3.2. Kodonoptimalizálás a fordítási pontosság érdekében
Az előző szakaszban bemutatott kodonoptimalizálás a transzlációs pontosság számba vételének hiányától szenved [36,37]. Vegyük például az E. coli {XE "E. coli"} Asn kódokat AAC és AAU. Az AAC egy major kodon (amelyet a nagymértékben kifejeződő gének erősen használnak, és amelyet a legnagyobb mennyiségben előforduló izoakceptor tRNS {XE "tRNS: isoacceptor"}{XE "isoacceptor tRNS"} dekódol), míg az AAU egy ritkán használt minor kodon. A nagymértékben expresszálódó E. coli gének szinte kizárólag az AAC-t használják Asn kódolására, így azt lehet állítani, hogy az AAC túlzott használatát a transzlációs hatékonyságra irányuló szelekció vezérli. Az AAC és az AAU azonban a félreolvasási arányban is különbözik, különösen a tRNALys által, amelynek ideális esetben csak az AAA és AAG kodonokat kellene dekódolnia, de az AAC-t és az AAU-t félreolvassa, ami az Asn-t Lys-re cseréli. Ez a hibás olvasási hibaarány az AAU esetében hatszor nagyobb, mint az AAC esetében, és a hibaarány következetesen fennmarad különböző kísérleti beállítások mellett, például Asn-éheztetett és nem éheztetett körülmények között [38], vagy a transzláció gátlására használt Streptomicinnel [39]. Ezért a nagymértékben expresszált E. coli gének túlzott AAC-használatát vagy a fokozott transzlációs hatékonyságra vagy a fokozott transzlációs pontosságra irányuló szelekció, vagy mindkettő okozhatja.
Akashi [37] kísérletet tett a szelekció transzlációs hatékonyságra és pontosságra gyakorolt hatásának szétválasztására. Az aminosavhelyeket konzervált (feltételezhetően funkcionálisan fontos) és változó (feltételezhetően korlátozott jelentőségű) helyekre osztályozta. Ha a kodonadaptáció a transzlációs hatékonyságra irányuló szelekciónak köszönhető, akkor a génben lévő összes kodonnak hasonló szelekciónak kell alávetnie magát, függetlenül attól, hogy a kodon funkcionálisan fontos vagy jelentéktelen helyen található. Ezzel szemben, ha a kodonadaptációt a fordítási pontosságra irányuló szelekció vezérli, akkor a szelekció erősebb a funkcionálisan fontos helyeken, mint a funkcionálisan nem fontos helyeken. Ez azt jelenti, hogy a funkcionálisan fontos kodonhelyeken nagyobb a szelekciós hatás, mint a funkcionálisan nem fontos kodonhelyeken. Nagyobb kodonadaptációt talált konzervált aminosavhelyeken, mint változó aminosavhelyeken. Ez összhangban van azzal a következtetésével, hogy a konzervált és a variábilis helyek közötti különbség a pontosságra való szelekció eredménye.
A megfigyelés azonban összhangban van a transzlációs hatékonyságra irányuló szelekcióval is. Vegyük például a lizin kodonokat (AAA és AAG) és a glutamát kodonokat (GAA és GAG). Tegyük fel, hogy az AAA kodon hatékonyabban dekódolható, mint az AAG, és a GAG hatékonyabban dekódolható, mint a GAA. Tegyük fel továbbá, hogy egy erősen expresszálódó ősi gén erős kodonadaptációt fejlesztett ki, amelyben a lizin főként AAA-val, a glutamát pedig főként GAG-gal kódolt. Most néhány lizinhelyen előfordulhat, hogy nem szinonim szubsztitúció történik {XE "nem szinonim szubsztitúció"} AAA-ról GAA-ra. Ezeket a helyeket most változó (funkcionálisan nem fontos) helyeknek nevezzük, és egy kisebb kodon GAA foglalja el őket. Ez a "gyenge kodonadaptáció" és a változó (funkcionálisan nem fontos) helyek közötti összefüggést eredményezné, amelynek kevés köze van a fordítási pontossághoz. Akashi [37] tárgyalta ezt a problémát, de nem adott végleges megoldást.
hogy empirikusan jellemezzük az egyes szinonim kodonok dekódolási hibaarányát a vázizomsejtekben, és kiválasztjuk a legalacsonyabb hibaarányú kodont. Az E. coli-ban lefordítandó mRNS esetében az AAU átkódolása AAC-ra növelné a pontosságot, mivel az AAC hibás olvasási hibája hatszor kisebb, mint az AAU-é. Egy alternatíva ismét a funkcionálisan fontos és nagymértékben kifejeződő gének, például a riboszómális fehérjék vagy a vázizomsejtekben nagymértékben kifejeződő gének kodonhasználatának követése a 2. táblázatban.
Fontos, hogy a vakcina mRNS-ét pontosan lefordítsák, mert a rossz aminosavak téves beépülése összezavarná immunrendszerünket a célpont felismerésében.
3.3. A transzláció beindításának jele
Az optimális kodonhasználat a hatékony transzlációs indítás nélkül nem növeli a fehérjetermelést [10,11], mivel a transzláció indítása gyakran a sebességet korlátozó lépés. A hatékony transzlációs iniciáció az emlősöknél elsősorban két tényezőtől függ [40,41]: (1) a Kozak-konszenzus [42,43,44], és (2) a másodlagos szerkezet, amely beágyazhatja a Kozak-konszenzust, hogy elfedje ezeket az alapvető transzlációs iniciációs jeleket [9,41,45]. Ezek a tényezők hozzájárulnak ahhoz, hogy a riboszómák megfelelően helyezkedjenek el a startkódonoknál, hogy a transzláció iniciációjától az elongációig át tudjanak haladni. A start kodon jellegét nem veszem figyelembe, mint a transzláció megindításának hatékonyságához hozzájáruló lényeges tényezőt, mivel az emlősök génjeiben a start kodonok használata alig változik. Például a humán kromoszóma 1 (Chr01, NC_000001.11) 11 327 annotált fehérjekódoló génje és splice izoformája közül csak öt gén (OAZ3, FNDC5, FNDC5, RNF187 és WDR26) egyik izoformája rendelkezik nem-AUG startkodonnal.
3.3.1. Humán transzlációs iniciációs konszenzus
Az emlős génekre vonatkozó, a transzláció iniciálását elősegítő Kozak-konszenzus a GCCRCCAUGG [40,46], ahol az AUG a start kodon. Ennek a konszenzusnak a megerősítésére az 5. táblázatban az AUG startkódont határoló helyspecifikus nukleotidfrekvenciákat mutatom be az összes fehérjekódoló génre (beleértve az izoformákat is) a humán Chr01-ből. A kapcsolódó pozíciósúlymátrix [23,24,47,48], amely az intronok nukleotidfrekvenciáit (0,26398, 0,21777, 0,22622 és 0,29203 A, C, G és T esetében) használja háttérfrekvenciaként, a GCCRCCAUGG konszenzussal összhangban lévő helyspecifikus mintázatot mutat. Ez a mintázat összhangban van más emberi kromoszómák génjeivel, és erősebb a magasan expresszált géneknél, mint az alacsonyan expresszáltaknál.

A két mRNS-vakcina a GCCACCAUG kódot használta, de az AUG startkódon utáni kodont nem, két okból kifolyólag. Először is, míg a -3R (az 5. táblázat első oszlopának 4. helye) többször is bizonyítottan fokozza a transzláció megindulását, addig a +4G (az 5. táblázat 10. helye), valamint az utána lévő nukleotidok transzlációindításra gyakorolt hatása nem volt meggyőző [46,49,50,51]. A +4G túlsúlyát az aminosav-kényszerhipotézissel [52,53] a következőképpen magyarázták. A fehérjék mintegy 60%-ánál tapasztalható N-terminális metionin-kivágás (NME), amelyhez egy kis és nem poláros aminosavra, például alaninra és glicinre van szükség. Az alanint a GCN, a glicint pedig a GGN kódolja, ami a G nagy gyakoriságához vezet a +4 helyen. Kevés bizonyíték van arra, hogy a +4G és a következő nukleotidok hozzájárulnak a transzláció beindításához. Másodszor, a második aminosav a spike fehérjében fenilalanin, ami biztosítja, hogy az NME nem történik meg. Ha ezt GCG-re (alanint kódoló) cserélnénk, az NME kiszámíthatatlan változásokat eredményezne az S-fehérjében. Ezen okok miatt az első kodont nem vesszük figyelembe a Kozak konszenzusoptimalizálás során.
3.3.2. 5′-UTR és másodlagos szerkezet
A start kodont kísérő 5′-UTR két kulcsfontosságú funkciót lát el: az mRNS stabilizálását és a kis riboszóma alegység által a start kodon lokalizálásának megkönnyítését. Az 5′-UTR optimalizálására három stratégia létezik az mRNS-vakcina fejlesztésében. Az első az, hogy egyszerűen egy magasan expresszálódó emberi gén 5′-UTR-jét vesszük, mint például a humán α-globin gének 5′-UTR-jét. A második a SARS-2-S természetes mRNS-ének használata. Ez az első két stratégia feltételezi, hogy a természetes szelekció által végzett optimalizálás extrapolálható az izomsejtekben történő transzlációra. A harmadik a ligandok szisztematikus evolúciója exponenciális dúsítással (SELEX), amelyet a 3′-UTR optimalizálásához használtak [54], de adaptálható az 5′-UTR optimalizálására is. Egy világjárvány elleni vakcina tervezésénél a gyors fejlesztés a legfontosabb, ezért az első két megközelítés tűnik a legészszerűbbnek.
A BNT162b2 5′-UTR-jének tervezése az első megközelítést követte, a humán α-globin 5′-UTR-jének beépítésével (az 5′-UTR azonos a humán HBA1 és HBA2 között), a Kozak-konszenzus kisebb módosításával (2A. ábra). Amint azt az 5. táblázatban korábban bemutattuk, az optimális Kozak-konszenzus a GCCACCAUG, amely az eredeti ACCAUG helyettesítésére szolgál (2A. ábra). Ez természetesen következik a 3′-UTR tervezésének korábbi megközelítéseiből, amelyek a humán α-globinból és β-globinból származó stabilitást biztosító szabályozó elemek beépítésével tervezték meg a 3′-UTR-t [13]. A magasan expresszálódó emberi gén 5′-UTR-jének használatának további előnye, hogy az ilyen 5′-UTR-ek szinte kivétel nélkül nem tartalmaznak olyan upstream AUG-ot, amely zavarhatja a transzláció beindulását.

Amint azt a transzláció iniciációjával kapcsolatos korábbi vizsgálatok élesztőben [55] és egérsejtvonalakban [56] kimutatták, a start kodon előtti vagy azt kísérő stabil másodlagos szerkezet az 5′-UTR-ben csökkenti a fehérje termelődését. A start kodon utáni szekunder struktúra azonban inkább fokozza a start kodonok eukarióta riboszómák általi felismerését [57]. A BNT162b2-nek kevés másodlagos szerkezete van a start kodont szegélyező, az 55-57. középső ablakhelyeken elhelyezkedő start kodon mellett (2B ábra). A Moderna mRNS-1273 azonban sajátos módon rendelkezik a start kodont keretező másodlagos szerkezettel (2B. ábra), amely a 2C. ábrán látható. Az mRNS-1273 esetében a 40 nukleotidra vonatkozó MFE a start kodonnal a közepén -12,3 37 °C-on.
A Moderna számos alternatív 5′-UTR és 3′-UTR szekvenciát fejlesztett ki, amelyek két szabadalomban is szerepelnek [18,19]. A Moderna mRNS-1273 5′-UTR-je a V1-UTR (SEQ ID NO 181) [18]. Két elemből áll. Az első elem a SEQ ID NO 1 egy 16 120 szekvenciát tartalmazó listán szerepel egy szabadalomban [19]. Ezt az elemet egy GC-gazdag második elem követi CCCCGGCGCC [18], közvetlenül a Kozak konszenzus ACCAUG előtt. Ez a GC-gazdag elem és az általa létrehozott másodlagos szerkezet (2C ábra) növelheti az mRNS stabilitását és a transzlációs pontosságot a szivárgó leolvasás csökkentésével [18].
Nem ismert, hogy egy ilyen másodlagos szerkezet az mRNS-1273 5′-UTR-ében (2C ábra) akadályozná-e a start kodonnak a cap-függő szkennelését, és kevésbé hatékony transzlációt eredményezne. A BNT162b2-nek a humán α-globin gén 5′-UTR-jéből származó 5′-UTR-je (2. ábra) azonban úgy tűnik, hogy ebből a szempontból jobb, mint az mRNS-1273. Mint már említettem, az mRNS-1273-nak a kodonoptimalizálással is van problémája (pl. a CGG túlzott használata, amely nem optimális kodon). Ezek a tényezők együttesen negatívan befolyásolhatják az mRNS-1273 transzlációs hatékonyságát, és hozzájárulhatnak ahhoz, hogy a Moderna vakcinának a Pfizer/BioNTech vakcinához (30 μg/dózis) képest magas dózisra van szüksége (100 μg/dózis). Ha egy mRNS-t nem fordítanak le hatékonyan, akkor több mRNS-re van szükség a kódolt tüskefehérje azonos mennyiségének előállításához.
Óvatosan kell eljárni a 2C) ábrán látható másodlagos szerkezetre vonatkozó fenti értelmezéssel). Néhány magasan expresszálódónak ismert génnek hasonló MFE-értékei vannak a középen lévő start kodonnal rendelkező 40 nukleotidban. Például a riboszómális fehérje gének (beleértve a különböző izoformákat is) 8,6%-ának MFE értékei a 40 nt-os ablakband egyenlőek vagy kisebbek, mint -12,3 (3. ábra). Más szóval, a riboszómális fehérje gének ezen 8,6%-ának másodlagos szerkezete ugyanolyan stabil vagy még stabilabb, mint az mRNS-1723-é. Ezért az mRNS-1273 másodlagos szerkezete (2. ábra) nem feltétlenül jelent alacsony transzlációs hatékonyságot.

Számos nagymértékben expresszálódó emberi gén 5′-UTR-je hasonló másodlagos szerkezettel rendelkezik. Például a humán α-globin gén 40 nt-os ablakának (20 nt a start kodon AUG előtt és 20 nt a CDS 5′ végében) MFE értéke -11,1, ami csak kissé gyengébb szekunder szerkezetet jelez az mRNS-1273 megfelelő régiójához képest, amelynek MFE értéke -12,3 volt. Az emlősök magasan expresszálódó génjei azonban jellemzően sokkal GC-gazdagabbak a start kodont szegélyező szekvenciákban, mint az alacsonyan expresszálódó gének, így az előbbiek általában stabilabb másodlagos szerkezettel rendelkeznek, mint az utóbbiak. Ez a GC-gazdagságbeli különbség a magasan és alacsonyan expresszálódó gének között mind a kódoló szekvencia előtti 5′-UTR-re, mind a kódoló szekvencia utáni 3′-UTR-re igaz.
Számos nagymértékben expresszálódó emberi gén 5′-UTR-je hasonló másodlagos szerkezettel rendelkezik. Például a humán α-globin gén 40 nt-os ablakának (20 nt a start kodon AUG előtt és 20 nt a CDS 5′ végében) MFE értéke -11,1, ami csak kissé gyengébb szekunder szerkezetet jelez az mRNS-1273 megfelelő régiójához képest, amelynek MFE értéke -12,3 volt. Az emlősök magasan expresszálódó génjei azonban jellemzően sokkal GC-gazdagabbak a start kodont szegélyező szekvenciákban, mint az alacsonyan expresszálódó gének, így az előbbiek általában stabilabb másodlagos szerkezettel rendelkeznek, mint az utóbbiak. Ez a GC-gazdagságbeli különbség a magasan és alacsonyan expresszálódó gének között mind a kódoló szekvencia előtti 5′-UTR-re, mind a kódoló szekvencia utáni 3′-UTR-re igaz.

A SARS-CoV-2 főként a TRS2 és TRS3 -t használja. Az Orf7b és az orf10 a CDS-ük előtt TRS1-et tartalmaz, de alig van kimutatható szubgenomiális transzkriptumuk. Ezzel szemben más ORF-ek TRS2-t és TRS3-at használnak, és számos szubgenomiális transzkriptummal rendelkeznek a transzkriptomiális szekvenciákban [26,61]. Az NCBI-ból letöltött SRR11886744.sra transzkriptomikai fájlban 46 olyan transzkriptomikai olvasatot találtam, amelyek szinte a 4C ábrán látható szekvencia teljes hosszának megfelelnek. Ez a transzkriptomikai fájl megfelel a GC-26/66 jó minőségű, hosszú leolvasásokat tartalmazó mintának [26]. Ez az információ hasznos lehet egy olyan terápiás szer kifejlesztésében, amely a TRS3-at célozza, hogy megszakítsa ezt a szakaszos transzkripciót.
Megjegyzendő, hogy a természetes SARS-2-S nem rendelkezik a GCCACCAUG optimális Kozak-konszenzussal, bár rendelkezik -3A-val, amelyet különösen fontosnak ítéltek a transzláció iniciálásához [40]. Ez jellemző az emlősvírusok génjeire. Ahogy Nakamoto [62] is érvelt, amikor a transzlációindító szekvencia nem rendelkezik jó Kozak-konszenzussal az emlősökben (vagy Shine-Dalgarno szekvenciával a prokariótákban), a szekunder szerkezet által nem akadályozott start kodon kulcsfontosságúvá válik a hatékony transzlációindításhoz. A 4C. ábrán látható szekvencia nem rendelkezik a start kodont beágyazó szekunder struktúrával.
3.4. Fordítás befejező jel
A legtöbb prokarióta fajjal ellentétben, ahol két felszabadító faktor van (az RF1 dekódolja az UAA-t és az UAG-t, az RF2 pedig az UAA-t és az UGA-t), az eukarióta felszabadító faktor eRF1 mindhárom stop kodont felismeri [63,64]. Ez a tény azonban nem jelenti azt, hogy a három stop kodon egyformán optimális az eukariótákban. Valójában több bizonyíték is arra utal, hogy a három stop kodon között nagy különbség van a befejezés hatékonyságában és pontosságában. A Pfizer/BioNTech BNT162b2 mRNS-e két egymást követő UGA stop kodont tartalmaz. A Moderna mRNS-1273 mindhárom különböző UGAUAAUAG stop kodont használja. Ezek az optimális elrendezések?
3.4.1. Hatékonyság és pontosság a transzlációs terminációban
A termináció hatékonyságát az egységnyi idő alatt dekódolt stop kodonok számával, a termináció pontosságát pedig a helyesen dekódolt stop kodonok arányával mérjük, szemben a közel-kognitív tRNS-ek (nc-tRNS-ek) általi téves olvasással. A hatékonyságot kísérletileg gyakran egy tRNS-hez kötött modellpeptid felszabadulásával mérik a riboszóma-komplexen eRF jelenlétében [63,64,65]. Ezek a vizsgálatok arra utalnak, hogy az UAA (különösen az UAAA) hatékonyabb, mint más stopjelek.
A pontosságot működési szempontból a stop kodonok nc-tRNS-ek általi félreolvasásának gyakoriságával (leaky termination) mérik. A stop kodonok tRNS-ek általi félreolvasásának arányát több baktériumfaj esetében is mérték. Az UGA a három közül a legkönnyebben átjárható, az átolvasási gyakorisága legalább 10-2-10-3 a Salmonella typhimurium [66] és az E. coli {XE "E. coli"} esetében. [67,68]. Az UAG és UAA stop kodonok esetében is előfordul az áttörés baktériumokban [69,70], de kisebb arányban. A természetes UAG áttörés gyakorisága többnyire 1,1 × 10-4 és 7 × 10-3 közötti tartományban van, a downstream nukleotidok jellegétől függően [70,71,72,73]. Az UAA átolvasása 9 × 10-4 és 1 × 10-5 közötti frekvenciával történik [70]. A különböző baktériumfajok nagymértékben expresszálódó génjei erősen preferálják az UAA kodonokat még az erősen GC-gazdag genomokban is, a stop kodonok túlnyomó többsége UGA [74,75].
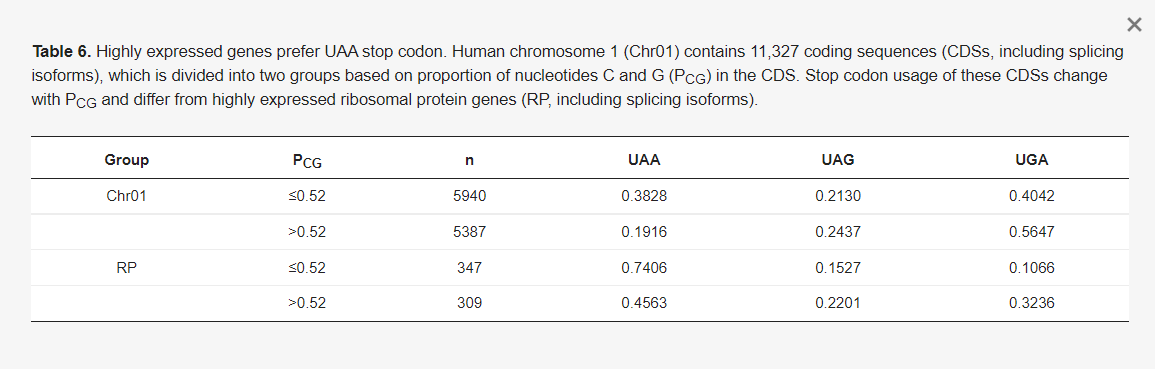
Különböző empirikus bizonyítékok azt sugallják, hogy az UAA a legjobb stop kodon az eukariótákban. Először is, az eRF1-hez való kötődési affinitás az UAA > UGA és UAG sorrendben van [63,64], ami arra utal, hogy az UAA hatékonyabb stop kodon, mint a másik kettő. Másodszor, az élesztőben a magasan expresszálódó gének az UAA stop kodonokat részesítik előnyben [76]. A 6. táblázatban szereplő empirikus adatok azt mutatják, hogy a magasan expresszálódó emlős gének szintén az UAA stop kodonokat részesítik előnyben. Ezt szemlélteti a humán kromoszóma 1 (Chr01) összes kódoló szekvenciájának (CDS) és a humán riboszómális fehérje géneknek (RP), amelyekről ismert, hogy magasan expresszálódnak, szembeállítása. A Chr01-en található összesen 11 327 CDS-t a GC-tartalom (PCG: a C és G aránya az egyes CDS-ekben) alapján két csoportra osztottuk. Az alacsony CG tartalmú csoportba tartozó CDS-ek PCG-je 0,3 és 0,52 között van, a magas CG tartalmú csoportba tartozó CDS-ek PCG-je pedig 0,52 és 0,8 között van (6. táblázat). Az alacsony-CG csoportban gyakrabban használják az UAA-t, mint a magas GC csoportban, ami a mutációs torzítás miatt várható. A Chr01 és az RP azonban drámaian különbözik az UAA használatában. A Chr01-ben mind az alacsony, mind a magas GC csoportban az UGA a leggyakoribb stop kodon. Ezzel szemben az RP gének az UAA stop kodont részesítik előnyben (6. táblázat). Ebben az összefüggésben a Pfizer/BioNTech BNT162b2-ben az UGAUGA és a Moderna mRNS-1273-ban az UGAUAAUAG terminációs jel nem biztos, hogy optimális választás.
Azt gondolhatnánk, hogy a két vakcina mRNS-ben egymás után található stop kodonok hibabiztos mechanizmust nyújtanak, tekintve az átolvasást, amelyet nemcsak az élesztőgének, hanem az emlősök génjeinek fordításában is megfigyeltek [77,78,79]. Például a humán MDH1-nek van egy UGA stop kodonja, amelyet gyakran Trp (UGG által kódolt) vagy Arg (CGA és más szinonim kodonok által kódolt) kódként fordítanak le, ami egy kiterjesztett fehérjét eredményez, frameshifting nélkül [77]. Egy további in-frame stop kodon várhatóan megakadályozza az ilyen nem kívánt, potenciálisan káros hatású fehérjék termelődését [80]. Prokariótákban azonban sok esetben az UGA stop kodonok téves olvasása +1 frameshift-tel jár [81,82,83,84]. Ilyen frameshifting eukariótákban is előfordul, például az emlős AZ1 gén UGAU stop kodon-kontextusával [85], ahol az UGA a stop kodon, amelyet egy U követ a +4 helyen. Az AZ1 mRNS fordításában a stopkódon első U-ját gyakran kihagyják, ha a poliaminok koncentrációja magas, ami azt eredményezi, hogy a riboszóma a GAU-t olvassa következő kodonként [85]. Egy ilyen +1-es kereteltolódás esetén a keretben lévő stoppkódon nem szolgálhat hibabiztos mechanizmusként. Az UGA rossz választás stop kodonnak, és az UGAU a Pfizer/BioNTech és Moderna mRNS-vakcinákban még rosszabb lehet.
3.4.2. Tetranukleotid terminációs jel
Már régóta felismerték, hogy a transzlációs terminációs jel több, mint egy stop kodon [86,87], ami a korai javaslathoz vezetett a tetranukleotid stop jelre, beleértve a downstream +4 helyet is [63,65,87,88]. Az előző fejezetben már utaltunk a +4U és a +1 frameshift összefüggésére. Az emlős eRF1 legújabb szerkezeti vizsgálatai [89,90,91] mechanisztikus magyarázatot adtak erre a tetranukleotid stop jelre. Az eRF1-ben lévő NIKS-motívumban lévő lizin (K) kölcsönhatásba lép a stopkódon első helyén lévő U nukleotiddal, hogy konformációs torzulást idézzen elő, így ahelyett, hogy a stop kodon három nukleotidja foglalná el az A helyet, négy nukleotid (stopkódon plusz a +4 hely) szorul be az A helyre, az eRF1 G626 glicinje pedig a +4 hely közvetlen fizikai közelségében van.
A nem poláros aminosavak jellemzően nem képeznek hidrogénkötéseket a nukleinsavakban lévő bázisokkal. A glicin azonban kivételt képez. A fehérjében lévő aminosavak és a DNS-ben lévő bázisok között megfigyelt kapcsolat alapján a glicin sokkal kedvezőbb kölcsönhatásba lép a purinnal, mint a pirimidinnel [92]. Ha ezt a megfigyelést a DNS-ről az RNS-re extrapolálhatjuk, akkor azt jósolnánk, hogy a G626 a +4 helyen lévő purint részesíti előnyben, azaz a +4 helyen lévő purin nagyobb valószínűséggel erősíti a leállási jelet a G626-tal való kölcsönhatás révén az eRF1-ben, mint egy pirimidin. Hasonló előrejelzéseket lehet tenni a +5 vagy +6 helyekre vonatkozóan is, amelyek azonban a +4 helynél sokkal kevésbé következetes hatást gyakorolnak a transzlációs terminációra [65].
Fontos a +4-es helyre vonatkozó jóslatot tesztelni, mert ha a +4-es helyen lévő purin valóban kedvez a transzlációs terminációnak, akkor a két mRNS-vakcina, +4U-val (valójában +4Ψ), nem rendelkezik jó terminációs jellel. A jóslatot ismét tesztelhetjük, ha a humán Chr01 gének RP génjei között a +4 helyen lévő nukleotidhasználatot állítottuk szembe egymással. Az olyan funkcionálisan fontos és nagymértékben kifejeződő gének, mint az RP gének, várhatóan erős terminációs jeleket fejlesztenek. Ha a purin a +4 helyen előnyös, akkor az RP géneknek átlagosan nagyobb valószínűséggel kell +4R-t tartalmazniuk, mint a Chr01 géneknek. A Chr01 CDS-ek (beleértve az izoformákat is) 11 327 annotált CDS-t tartalmaznak, következésképpen 11 327 stop kodont. Ezek között 3306 UAA, 2578 UAG és 5443 stop kodon található (7. táblázat). Az UAA, UAG és UGA végződésű CDS-eknél a három stop kodon mindegyikéhez tartozó +4A, +4C, +4G és +4U százalékos aránya látható (7. táblázat). Ezek a százalékos értékek értelmet nyernek, ha összehasonlítjuk a magasan expresszálódó RP-gének 760 annotált CDS-ére (beleértve az izoformákat is) vonatkozó értékekkel. A hipotézis, miszerint a magasan expresszált gének a +4R-t részesítik előnyben, erősen alátámasztott, ami mindhárom stop stop kodon esetében konzisztens (7. táblázat). A funkcionálisan fontos és magasan expresszált RP gének sokkal inkább használják a +4R-t, mint egy átlagos gén, amelyet a Chr01 gének képviselnek. A különbség a likelihood ratio teszttel vizsgálva rendkívül szignifikáns (likelihood ratio chi-négyzet = 931,0514, DF = 17, p <>

A +4R erős preferenciája a magasan expresszált RP génekben (7. táblázat) összhangban van más empirikus bizonyítékokkal. A +4C-vel ellátott UGA stopkódon a leghajlamosabb az átolvasásra az emlősök génjeiben, különösen az UGA CUA kontextusában [77,78]. Mind az UGAC, mind az UGAU gyakori szelenocisztein beépüléshez vezet [65], ami arra utal, hogy az UGA mint +4Y-vel rendelkező stop kodon rosszul dekódolható. Emlősök génjeiben a +4 hely hatása mindhárom különböző stop kodon között konzisztens a kísérleti vizsgálatokban, a termináció hatékonysága UAAR >> UAAY, UAGR >> UAGY és UGAR >> UGAY mind in vitro, mind in vivo [65]. Az UGAC terminációs hatékonysága például <20%-a az="" uaaa-nak="" [65].="" a="" 7.="" táblázat="" azt="" sugallja,="" hogy="" ezek="" a="" konkrét="" szekvencia-konstrukciókkal="" és="" transzlációs="" rendszerekkel="" kapott="" korai="" kísérleti="" eredmények="" általánosak="" és="" valósak.="" röviden,="" a="" két="" mrns-vakcinában="" az="" optimális="" stopjelnek="" az="" ugau/uagu/uaau="" helyett="" az="" uaaa-nak="" kell="">
A fenti érvelés egyik fenntartása az U N1-metil-pszeudouridinre (Ψ) való cseréjére vonatkozik a két vakcina mRNS-ben. Annak érdekében, hogy a gazdasejtek ne támadják idegen RNS-ként az exogén vakcina mRNS-t [13,58], az mRNS-vakcinákban az összes uridint Ψ-re cseréltük [2,14]. Ezért a Pfizer/BioNTech vakcinájában az UGAUGA helyett ΨGAΨGA, a Moderna vakcinájában pedig UGAUAAUAG helyett ΨGAΨAAΨAG a stopjelek. Mivel a Ψ a bázispárosodásban promiszkuzívabb, mint az U, és mind A-val, mind G-vel, és kisebb mértékben C-vel és U-val is párosulhat [16], a stop kodonok hajlamosabbak a tRNS-ek általi félreolvasásra [17]. Ez az oka annak, hogy mindkét mRNS-vakcina egymást követő stop kodonokat használ hibabiztos mechanizmusként, abban a reményben, hogy az első stop kodon sikertelensége esetén nem következik be kereteltolódás. Az UGAU azonban köztudottan +1 frameshiftinget okoz. Ésszerűen következtethetünk arra, hogy a ΨGAΨ is ugyanez lehet. Korábban már említettem, hogy az emlős AZ1 gén UGAU stopkódon-kontextussal hajlamos a poliaminok által kiváltott +1 frameshiftingre [85]. Az ilyen +1 frameshifting meghiúsítja a többszörös stop kodonok mint hibabiztos mechanizmus célját.
5. Az mRNS-vakcinák 3′-UTR-je
Korábban már említettem az 5′-UTR és a 3′-UTR optimalizálásának különböző megközelítéseit. Elegendő idő esetén a ligandumok exponenciális dúsítással történő szisztematikus evolúciója (SELEX) [54] lehet az előnyben részesített módszer. Vészhelyzetben azonban a természetből való kölcsönzés alternatív megközelítése hatékonyabb lehet. A Pfizer/BioNTech vakcina mRNS-ének 5′-UTR-je egy emberi α-globin gén 5′-UTR-jét tartalmazza (2A ábra), ami azért ésszerű, mert az α-globin mRNS-ek nagyon hatékonyan transzlálódnak. Ugyanezt a természetből kölcsönzött megközelítést alkalmazták a terápiás mRNS-ek 3′-UTR-jének tervezésénél is, pl. a humán α-globin és β-globin gének stabilitást szabályozó elemeinek beépítésével [13]. Ezek a stabilitást szabályozó elemek gyakran RNS-fehérje komplexeket alkotnak az mRNS stabilizálása érdekében [93,94,95,96,97]. A globin gének 5′-UTR és 3′-UTR elemei más mRNS-ekhez ligálva stabilitást kölcsönözhetnek ezeknek az mRNS-eknek [54,98,99]. A Moderna mRNS-1273 "beillesztette" a humán α-globin gén (HBA1) 110 nt-os 3′-UTR-jét az utolsó stop kodon és egy poli(A) farok közé. 3′-UTR tervezése a Pfizer/BioNTech mRNS-vakcina esetében a SELEX és a természetből való kölcsönzés kombinációja. A cél olyan, a természetben előforduló RNS-szegmensek megtalálása, amelyek jobban teljesítenek, mint a humán β-globin mRNS 3′-UTR-je [54]. A SELEX optimalizációs protokoll segítségével két RNS-szegmens teljesít jobban a többi alternatívánál [54]. Az egyik a humán mitokondriális 12S rRNS-ből (mtRNR1), a másik szegmens pedig a humán AES/TLE5 génből származik. Mivel e két RNS-szegmensről derült ki, hogy a miRNS-ek számára a legkevesebb prediktált kötőhelyet és a legmagasabb hibridizációs energiát tartalmazza [54], az AES-szegmensbe két C→U mutációt vezettek be a kötési energia további növelése érdekében (MFE = -37-ről -39,3-ra 37 °C-on, DAMBE-ból végzett számításom). A Pfizer/BioNTech mRNS-vakcina esetében a két C→Ψ mutációt tartalmazó 136 nt hosszúságú AES-szegmenst közvetlenül a második stop kodont követő két trinukleotid után illesztették be. A 139 nt hosszúságú mtRNR1 szegmenst közvetlenül utána illesztették be. A természetből való kölcsönzés heurisztikus és empirikus megközelítése vészhelyzetben talán hatékonyabb, mint az alternatívák.
4. Következtetések
A két széles körben használt mRNS-vakcinát, az egyiket a Pfizer/BioNTech, a másikat a Moderna cégtől, a magasan expresszált emberi génekből való kölcsönzéssel optimalizálták. Azonban több nem megfelelő optimalizálás is van. Kiemeltem és illusztráltam az ilyen eseteket abban a reményben, hogy a koncepcionális keret megkönnyíti nemcsak a vakcinák, hanem más terápiás mRNS-ek tervezését is.



